非典疫情专家:SARS CoV是非自然方式产生

SARS于2002-11在广东突然发生,2003-07后,除实验室感染和广州之小爆发外,地球上再无病例。更令人惊异的是,广州爆发病例的临床与流行病学特点和之前流行的截然不同:症状轻无续发。而之后数月实验室感染病例却和2002〜2003年流行的完全一致。这在世界传染病流行史上史无前例。有学者认为蝠SARS样冠状病毒(Bt-SLCoV)Rp3(DQ071615)株为SARS CoV的共同祖先,但非直接祖先,且和SARS CoV的进化间隔尚有4.08年,继后相关专家不仅在内地和香港密集搜索,且在东南亚甚至几大洲寻找,终未见其直接祖先踪影。因此,虽然在高科技的今天人们力尽所能,而SARS CoV的起源和贮存宿主仍是个迷。Bt-SLCoVRp3株可能为SARS CoV的共同祖先,但非直接祖先,与SARS CoV的进化间隔尚有4.08年。而在动物传染病病毒由动物适应于人类的历史中,4年时间非常之短,短得可忽略不计。以HIV的起源为例,其源病毒为猴免疫缺陷病毒,虽在其宿主内进化速度相当快,但其传人并适应于HIV-1的贮存宿主黑猩猩,成为后者携带的SIV(SIVcpz),经历几百年,SIVcpz的最近共同祖先时间为1492年,然后又经几百年适应性进化,才以HIV-1的形式于1921〜1963年间分多次传人人群。所以,若在自然情况下,不可能在如此短的时间内,由Bt-SLCoV经适应性进化至食肉目动物再至人类。故只能经“非寻常进化(UE)”方式,很可能是“非自然”的方式(如基因改造技术)产生SARS CoV。为何SARS CoV会发生强烈的“逆向进化”?原因在于,其以“非寻常进化”方式、很可能“非自然”地引人果子狸和人群后,遭到了新宿主群体强大持续而为SARS CoV不能适应的压力,故病毒只能以“逆向进化”应对,尽快回到其“祖先状态”,但此种“祖先状态”仅能在蝠中生存,故离开人群是其惟一出路。

【作者简介】徐德忠,无锡人,先后于北京医科大学本科和第四军医大学流行病学专业研究生(1968年)毕业;现任第四军医大学军事预防医学学院军队流行病学教研室教授和INCLEN CEU主任、博士生导师,享受政府特殊津贴,在职专业技术二级。 为全国著名流行病学专家,全军学术带头人,曾任卫生部非典疫情分析专家组组长、中华临床流行病学会副主任委员,全军流行病学专业委员会主任委员与顾问等学术职务。主要从事病毒性肝炎、新发传染病、食管癌、冠心病与遥感流行病学研究。负责国家、军队、省级课题24项:其中国家自然科学基金重点题1项、863军队专题1项。获全军育才奖金奖,吴阶平医学研究二等奖,省部级科技进步奖一等奖2项、二等奖7项。培养研究生100余名,其中博士后3名,博士生50多名。被评为全国非典防治工作先进个人、全国防治非典型肺炎优秀科技工作者、总后优秀党员。在国内外发表科研论文320余篇。共主编或参编专著30部;主编8部:包括国际首本“航空流行病学(已再版)”、国内首本“分子流行病学”和英文专著“传染病理论流行病学,Theoretical Epidemiology of Infectious Diseases”。主编的“循证医学入门.临床科研方法与实例评价(第二版)”获“第八届全国高校出版社优秀畅销书”;副主编(主编为程天民院士)的“军事预防医学”获“第六届中国人民解放军图书奖”和“第二届中华优秀出版物图书奖”。2003年参与国家和军队“非典”的防控,正确分析、预测了我国我军的流行趋势,除向军委和国家卫生部领导汇报外,还完成中共中央办公厅和国务院办公厅“简报”24期、报告3个,央视焦点访谈2期、新闻专题6期,受到军委首长和卫生部领导高度评价。
早在2005-02美国科学促进会(AAAS)年会上,一位长期从事冠状病毒研究的美国微生物学专家Kathryn Holmes指出,世界不可能再次面临如两年前一种突发的严重急性呼吸综合征(severe acute respiratory syndrome,SARS;又称传染性非典型性肺炎或非典)的爆发;在人群中曾经传播的SARS冠状病毒(SARS CoV),可能仅存在于实验标本内[1]。此话一出,令世人震惊,尤其遭到我国多位院士和学者的强烈反驳[2-3]。
我国学者的反驳理所当然,因为SARS给我国人民的健康和国民经济造成了巨大损失,至今令人印象深刻。这场灾难实际上是世界性的,不仅使我国内地发病5327例,死亡349例,还波及29个国家和地区,共发生8098例,死亡774例。
1、有待解答的关键问题
对于上述争论,目前有两个问题摆在国际科学家和公众面前,有待解答:
1.1 为何与其他病毒不同,SARS CoV起源至今无解?
SARS流行至今已近10年,无数学者进行了全面深人的研究,但在SARS CoV起源上始终未有突破。有学者认为蝠SARS样冠状病毒(Bt-SLCoV)Rp3(DQ071615)株为SARS CoV的共同祖先,但非直接祖先,且和SARS CoV的进化间隔尚有4.08年,继后相关专家不仅在内地和香港密集搜索,且在东南亚甚至几大洲寻找,终未见其直接祖先踪影。因此,虽然在高科技的今天人们力尽所能,而SARS CoV的起源和贮存宿主仍是个迷[4-6]。相反,2009-04新发现的新甲型流感(H1N1)病毒在全球流行,学者们很快发现,其由8种基因组成,主要来自4种谱系,和北美流行的三重配体猪型H1N1病毒尤其接近,在流行开始2个月内即被研究得如此清楚[7-8]。
1.2 为何与其他病毒不同,SARS在2004-01流行后,除实验感染外再无病例?
在2003-07后,除实验室感染和2003-12/2004-01广州4名轻型病例外,SARS在人群中再无病例发生,动物中也未见感染。与此相反,近年来,其他由动物和(或)人源性新发传染病,如获得性免疫缺陷综合征(acquiredimmunodeficiencysyndrome,AIDS),多型病毒性肝炎,上世纪90年代中期爆发的亨德拉病毒(Hendravirus,HV)病和尼巴病毒(Nipahvirus,NV)病以及2009年的甲型流感(H1N1)仍在发病地区甚至更广泛地域不断流行[9-10]。
2、现在自然界和人群中已不存在 SARS CoV 及其消失的原因
欲回答上述两个问题,必须在SARS CoV的起源和进化研究方面有突破传统的新思路和新方法。为何此领域的研究久久停滞不前,影响因素很多,但主要原因是,虽然国内外学者对SARS CoV基因结构和功能及其变异、进化及其机制等进行了大量研究,取得许多成果[4-6];但研究者大多来自分子生物学、微生物学、生物信息学等基础学科,多从微观角度研究问题,多从一个侧面寻求答案,却未从宏观角度去探讨,未从相反角度去思索:自然界是否可能没有出现过SARS CoV的直接祖先,自然界是否根本不存在SARS CoV的贮存宿主?我们在此从全新之视角,深人研究并综合分析SARS爆发以来国际上的主要工作和相关文献,结合流行病学和生物进化理论,得出结论:自然界根本不存在SARSCcV直接祖先和贮存宿主,故流行后,SARS CoV即从人群和自然界消失。其主要理由如下:
2.1 SARS的流行进程不符合迄今世界上传染病流行的自然史
SARS于2002-11在广东突然发生,2003-07后,除实验室感染和广州之小爆发外,地球上再无病例。更令人惊异的是,广州爆发病例的临床与流行病学特点和之前流行的截然不同:症状轻无续发。而之后数月实验室感染病例却和2002〜2003年流行的完全一致。这在世界传染病流行史上史无前例。早在SARS流行刚结束时,胡锦涛总书记阐述了对人类传染病流行史的认识,明确指出:“人类传染病史告诉我们,任何重大的传染病,都不会在一次发作后就销声匿迹。”然而SARS却相反。
可见,SARS的流行自然史有十分异常之处!其原因是什么?现在我们可指出,最重要原因是:自然界根本不存在SARS CoV的直接祖先,其是以“非寻常进化”方式,很可能是“非自然”地引人人群的,故不能遵循正常的流行规律。
2.2 SARS CoV的系统发育非同一般,其存在快速而明显的“逆向进化”
“逆向进化”被定义为“再取得祖先状态”,是进化过程的组成部分,在生物界包括微生物界普遍存在。在自然状态下的进化长河中,“逆向进化”可能主要是“顺向进化”之补充与协调,为进化主流之曲折与迂回。然而,SARS CoV的“逆向进化”却出现早、表现多、持续长、力量大。流行早期即丢失了和病毒宿主适应性相关的基因0RF8特征性29-nt,出现了和病毒毒力与传播力相关的受体结合位点重要氨基酸的“逆向进化”;2003~2004年广州爆发的SARS CoV和2002~2003年流行早期病毒之亲缘距离较晚期的更接近,等等。虽然,既往研究者对这些现象均进行了详细描述和分析,可是未用“逆向进化”理论作指导,或者尚未意识到“逆向进化”,因为目前国际上尚未将“逆向进化”理论用于人类疾病研究,因此,他们不能完全清楚地解释这些变异和进化现象,最后仅以“还需深人研究”结尾。
我们将这些研究结果重新审视和综合研究,提出了SARS CoV进化中存在明显的“逆向进化”,其主要结果不在此全面叙述,将另文发表。
同时,我们也做了一些具体研究[11-12],以GZ02(AY390556)为外群,构建了系统发育有根树(图1):将9条序列分为4组:PC03(SZ3SZ16),PC04(PC413、PC4136、PC4227),HP03(CUHK-Ag01、CUHK-Ag02),HP04(GZ301、GZ302)。结果发现,除CUHK-Ag01、CUHK-Ag02外,其它序列的位置及其相互关系Song等[4]研究结果一致。但是,我们增加的CUHK-Ag01、CUHK-Ag02位置及其2003-03上旬的TMRCA(TimeofMostRecentCommonAncestor,最近共同祖先时间)呈现出非常奇特的状况:TMRCA的计算结果不符合实际,因为分离到此2株病毒的患者3月中旬发病,但其病毒的祖先出现时间仅间隔10d,而此两株病毒和外群TMRCA又与实际相符。其原因可能是此两株病毒在流行后期,其选择压力、突变模式和进化方向正处在剧烈变动之中,使用目前国际上公认的计算公式和(或)建树方法,已不能正确地反映SARS CoV快速而明显的逆向进化实际。我们将图1和Song等[4]构建的无根树(图2)进行对比研究,发现SARS CoV的进化(距离)顺序为:2002~2003年流行晚期毒株—2002-2003年流行中期毒株^2002~2003年流行早期毒株—2003-2004年流行毒株,也即与正常的进化方向正好相反,这不仅支持了我们的上述观点,同时使我们认识到其在流行过程的进化中存在“逆向进化”。
然而,为何SARS CoV会发生强烈的“逆向进化”?原因在于,其以“非寻常进化”方式、很可能“非自然”地引人果子狸和人群后,遭到了新宿主群体强大持续而为SARS CoV不能适应的压力,故病毒只能以“逆向进化”应对,尽快回到其“祖先状态”,但此种“祖先状态”仅能在蝠中生存,故离开人群是其惟一出路。
现在应回答此问题:SARS CoV又是如何“非自然”地进人果子狸和人群的?
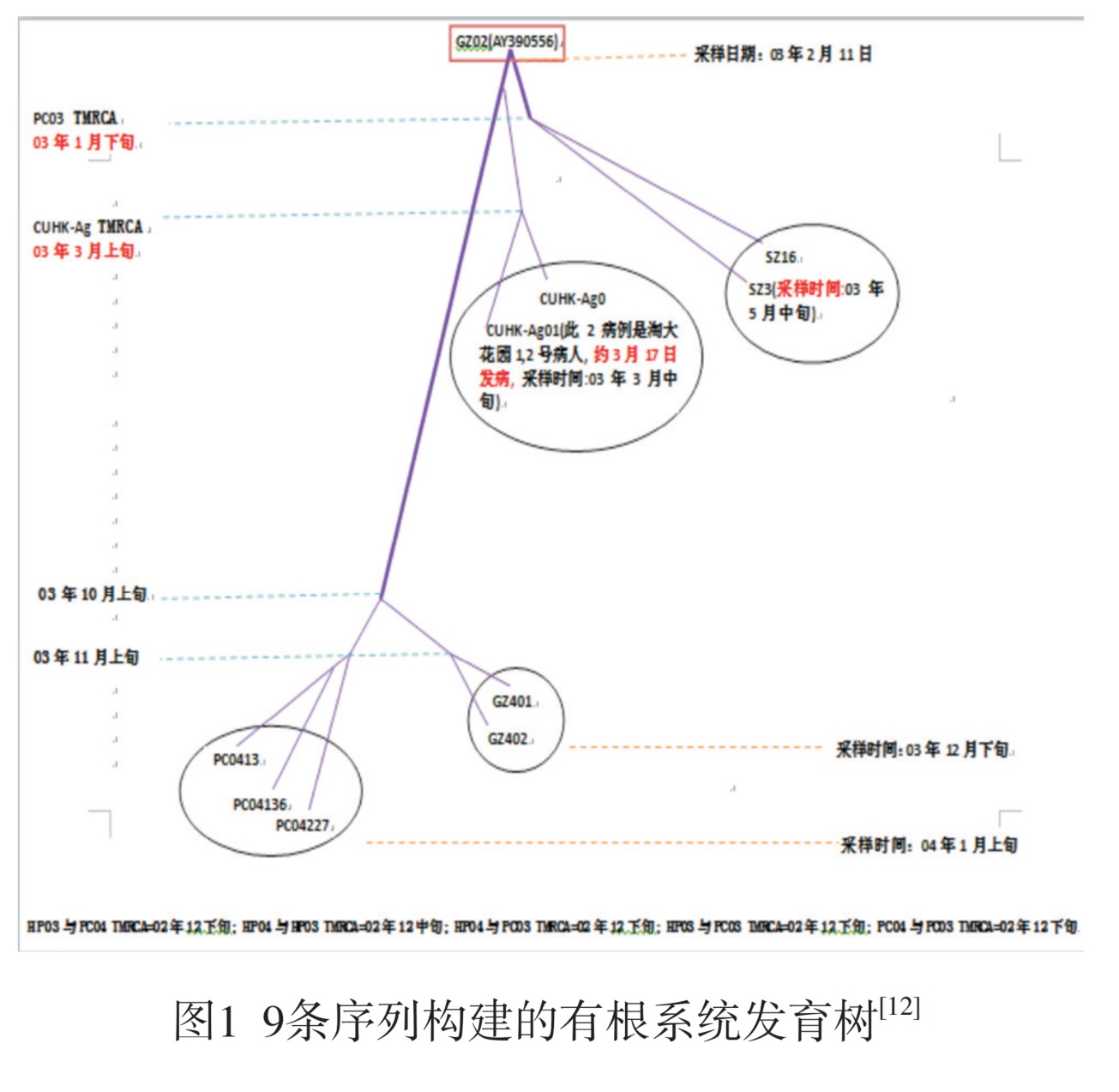
图1 9条序列构建的有根系统发育树[12]
1:TMRCA(Time of Most Recent Common Ancestor):最近共同祖先时间;2:左侧时间:为外群GZ02和各组之间TMRCA,或HP04与PC04之间TMRCA,或HP04或PC04 TMRCA:由水平虚线(……)指向发育树分枝节点;3:下端时间:为两个组之间TMRCA,除“PC04与PC03=03.1中”在图中未表示外,均由发育树树根处可见;4:TMRCA具体时间的缩写:如GZ02: 02、12上,表示2002-12上旬。

图2 Song构建的无根树[4,12]
2.3 SARS CoV“非自然”进入果子狸和人群的方式
已如前述,Bt-SLCoVRp3株可能为SARS CoV的共同祖先,但非直接祖先,与SARS CoV的进化间隔尚有4.08年。而在动物传染病病毒由动物适应于人类的历史中,4年时间非常之短,短得可忽略不计。以HIV的起源为例,其源病毒为猴免疫缺陷病毒(simianimmunodeficiencyvirus,SIV),虽在其宿主内进化速度相当快,但其传人并适应于HIV-1的贮存宿主黑猩猩(chimpanzees),成为后者携带的SIV(SIVcpz),经历几百年,SIVcpz的最近共同祖先时间为1492(1266〜1685)年,然后又经几百年适应性进化,才以HIV-1的形式于1921〜1963年间分多次传人人群[13]。
所以,若在自然情况下,不可能在如此短的时间内,由Bt-SLCoV经适应性进化至食肉目动物再至人类。故只能经“非寻常进化(UE)”方式,很可能是“非自然”的方式(如基因改造技术)产生SARS CoV。所以,我们对其起源和进化路线假设如下:蝠Bt-SLCoV(亲代3)——非寻常进化“UE-SLCoV1(非寻常进化-SARS样冠状病毒1)株”(亲代2)——UE-SLCoV2株(亲代1)——2002〜2003年果子狸SARS CoV株——2002〜2003年人SARS CoV株——2003〜2004年果子狸SARS CoV株——2003〜2004年人SARS CoV株 ——??——??——消失。故我们将SARS CoV谓之“过客病毒(passenger virus)”。
由此,本文在国际上首次阐明了SARS CoV的起源,并以多种学术证据判明:自然界根本不存在其直接祖先和贮存宿主,其也早已于流行后不久在自然界和人群中消失。在此应特别指出,上述假设的SARS CoV起源和进化路线,仅是大的框架,具体的环节尚待修改和完善。然而,应用此起源和进化路线的假设,即可合理解释本文开头提出的摆在科学家和公众面前的两个问题。
在此尚需说明一要点:将动物病毒改造成人类病毒的方法(如基因改造技术)成熟与否,在学术界有过争议。但是,现在应该毫无疑义了。因为,2012年国际顶级杂志于5、6月份发表了曾在2011年轰动世界自然科学界的两篇论文,用事实回答了这个问题[14-15]。然而,实际上在2000年甚至更早,国际上的各种资料和渠道透露,当时有些人正在研究甚至已经掌握了这些技术[16-17]。
最后,我们郑重建议我国卫生部:由于在SARS这场灾难中,我国损失最大,奉献最多,请正式向WHO和有关国际机构申请,组织特别专家委员会会同有关政府部门进行调查研究,实证现在自然界和人群中已不存在SARS CoV,并向世界宣布:迄今已经消灭了人类第二种传染病一—SARS。由此,不仅可使公众安心并从中吸取教训,而且可慰藉无私献身之医务卫生人员和其他英雄以及病故患者的在天之灵!
致谢:本项目组成员王波副教授、于晓寒硕士对本文也有贡献;本文得到本校陈景元、闫永平和夏洁来教授、张景霞高级实验师、张磊讲师的支持;笔者和陕西师范大学生命科学院黄原教授就生物进化的某些问题进行了讨论,受益匪浅;本校图书馆苏春萍副研究馆员和石建副研究馆员无私帮助收集文献;本校外语教研室樊家勇副教授对摘要的中译英给予重要指正。
【参考文献】
[1] BBC NEWS:http://news.bbc.co.uk/go/pr/fr/-/2/hi/health/4280253.stm,Published:2005/02/20 00:33:34 GMT.
[2] 文涛.SARS病毒是否巳在自然界彻底灭绝?[J].国外科技动态,2005,(3):28-31.
[3] 钟南山:“非典灭绝说”是种危险误导.http://scitech. people.com.cn/GB/25893/3196196.html.
[4] Song H,Tu C,Zhang G,et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and
human[J]. Proc Natl Acad Sci USA,2005,102(7): 2430-2435.
[5] Hon CC,Lam TY,Shi ZL,et al. Evidence of the recombinant origin of a bat severe acute respiratory syndrome (SARS)- like coronavirus and its implications on the direct ancestor of SARS coronavirus[J]. Virol,2008,82(4): 1819-1826.
[6] Balboni A, Battilani M, Prosperi S. The SARS-like coronaviruses: The role of bats and evolutionary relationships with SARS coronavirus[J]. New Microbiol,2012,35(1):1-16.
[7] Fraser C,Donnelly CA,Cauchemez S,et al. Pandemic potential of a strain of influenza A (H1N1): Early findings[J]. Science, 2009,324(5934):1557-1561.
[8] Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team. Emergence of a novel swine-origin influenza A (H1N1) virus in humans[J]. N Engl J Med, 2009, 360 (25):2605-2615.
[9] Hemelaar J, Gouws E, Ghys PD, et al. WHO-UNAIDS Network for HIV Isolation and Characterisation. Global trends in molecular epidemiology of HIV-1 during 2000- 2007[J]. AIDS, 2011, 25(5):679-689.
[10] Hasebe F, Thuy NT, Inoue S, et al. Serologic evidence of nipah virus infection in bats, Vietnam[J]. Emerg Infect Dis, 2012,18(3):536-537.
[11] 孙慧敏,唐晓凤,王波,等.SARS病毒和其他冠状病毒中性突变速率初步的比较研究[J].中华疾病控制杂志,2011, 15(12):1026-1030.
[12] 谭雅慧,孙慧敏,唐晓凤,等.SARS-CoV中性突变速 率和有根系统发育树再研究及其起源的新思考[J].中华 疾病控制杂志,2012, 16(10):860-863.
[13] Sharp PM, Hahn BH. The evolution of HIV-1 and the origin of AIDS[J]. Philos Trans R Soc Lond B Biol Sci, 2010,365(1552):2487-2494.
[14] Imai M, Watanabe T, Hatta M, et al. Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets[J]. Nature, 2012, 486(7403):420-428.
[15] Herfst S, Schrauwen EJ, Linster M, et al. Airborne transmission of influenza A/H5N1 virus between ferrets[J]. Science, 2012, 336(6088):1534-1541.
[16] 小征.基因武器即将问世[J].中国科技月报,1999,(6):56.
[17] 王家骥.谨防生物技术被滥用于发展危害人类的基因武器一斯德哥尔摩国际和平研究所1999年《世界军备和裁军年鉴》所载《生物技术和遗传工程发展的利弊》论文简介[J].国际问题研究,2000,(2):56-58.
【察网摘自《医学争鸣》2013年第1期,原标题《现在自然界和人群中已不存在非典病毒》】
相关阅读一:SARS_CoV非自然起源
作者:徐德忠 孙慧敏 谭雅慧 来源:《医学争鸣》2013年第4期
我们在国际上首次阐明了SARS CoV的起源,并判明自然界根本不存在其直接祖先和贮存宿主,因其流行后不久在自然界和人群中消失,故将SARS CoV谓之“过客病毒”(passenger virus)[1]。过去10年中,国内外无数学者对其基因结构及其变异与跨宿主进化进行了全面深入的研究,终未发现其真正起源[2-4]。我们认为,其原因主要是这些研究者多来自实验医学领域,未从宏观和微观结合上,未从同一疾病发展前后历史比较上和多病种相互比较上,以及未在创新思路上探讨SARS CoV起源的实质[1]。在此,我们认为,SARS CoV由Bt-SLCoV经“非寻常(如基因改造)进化(unnatural evolution,UE)”方式产生[1]。这实际上反映出人类已进入经基因改造技术等方式将某些低级野生动物病毒制成“新型人工病毒”并引起一国或多国乃至全球性流行的时代。故应引起科学家和公众的足够重视。为此,我们将从流行病学异乎寻常之分布和临床特征的明显反常角度,进一步详细阐明SARS CoV之非自然(基因改造)起源。
欲从流行病学上区分传染病的自然流行或非自然流行,也即区分是生物武器抑或自然界病原体致人或动物群体之流行,其策略为鉴别传染病的分布是否符合该病的自然史。我军已用和仍将用此基本原则对待敌方的生物战(以往谓细菌战)。因为传染病的分布(亦可称流行特征)为病原体及其进化,传染源即携带并能排出该病原体且使易感者受染之宿主、传播途径、易感人群三环节以及自然和社会环境二因素相互作用的结果,故若病原体起源不同,传播途径或释放病原体方式不同,必然致传染病的分布不同。流行病学专家可按透过现象看本质的哲学思维,判断病原体的真正起源和传播或释放方式。
1、广东2002~2003年3个流行期间病例分布、传播方式和临床表现差异显著且反常
2002-11-16我国佛山发现第1例SARS患者,至2003-01-31两个半月中发病194例,分散在6个市区,传播慢,症状较轻,病死率低;而其后2、3两个月共发病1037例,主要集中在广州(图1,2)[5-6],且为医院—家庭爆发模式,病情危重,出现较多超级传播者;并于2月下旬分别传至香港和北京,再扩散至东南亚和美欧诸国。值得一提的是,2003年4月中旬至5月初,每天SARS发病数达100~200例以上,但至2003-06-03即再无发病报告;当年7月后,除2003-12~2004-01广州爆发4例轻型病例和实验室感染病例外,近10年未见发病。此种流行规律和模式及其临床特征变化,在人类传染病史上尚无先例。
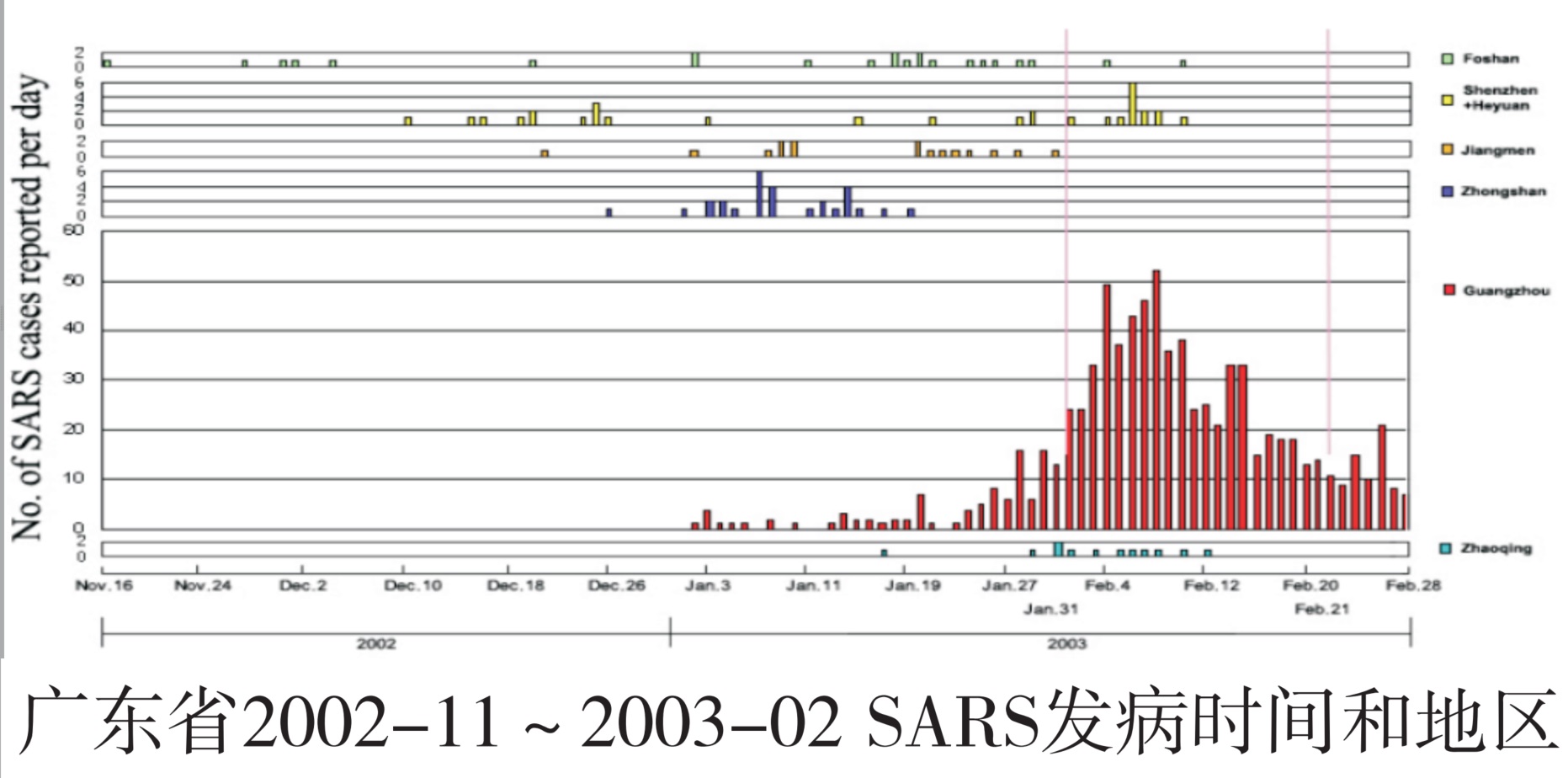
图1 广东省2002-11~2003-02SARS发病时间和地区分布[5]

图2 广东省SARS流行早期的地区分布[5]
更耐人寻味的是,SARS在广东省流行早中期的地区分布特点,《Science》杂志对此作了专门叙述:自2002-11-16第1例至2003-03-10东莞出现的指示病例期间(即中国内地3个流行阶段中的早期至晚期之初),广东省的病例均发生在广州西面和南面的深圳市,其余地区均无病例(图1,2);对于河源的发病,论文特别加以说明:其指示病例在深圳受染,当地无患者[5]。此种畸形地区分布发人深思:因为广州东面和北面城市在社会与自然环境等方面与西面和南面无明显差别。该文作者当时做如下解释:由于20世纪70年代初,珠江流域经济发展,居民饮食习惯改变,开始引进外来动物食品[5]。这似有一定道理,但却不能说明以下问题:①吃果子狸等习惯已数十年,但为何2002年才发生流行;②此习惯在广州周边均如此,为何流行早中期仅局限于广州的西面和南面;③能分离出SARS CoV的果子狸(图2)仅在广州和深圳两市的野生动物市场,而周边农村饲养场和其他省区均无[6-9]。因此,反映此种奇怪分布(外在表现)很可能的深层次原因(内部本质)是果子狸的受染和(或)受染的果子狸输入,即传染来源引入动物界和(或)人群是非自然的,简言之是人为的。此与我们之前指出的SARS CoV“逆向进化”过程相吻合[1]。
2、2003~2004年广州爆发流行的SARS临床特点及随后实验室感染与2002~2003年流行均截然不同
如上所述,2002~2003年SARS的流行分别在各国于2003年6~7月嘎然而止,但2003年底至次年初在广州爆发。4名病例相互无接触史呈散发状态,但其中3例在病前一个潜伏期内均有果子狸暴露史,1例病前有鼠暴露史[7]。令人惊异的是,该4名病例病情轻,均痊愈出院,发病到出院时间为22d左右,出院后预后好,且257名接触者(其中密切接触者113人)无一发病。Song等[2]指出,这次广州爆发病例分离出的SARS CoV,除了系统发生树和2002~2003年流行的有一定区别外,其ORF1a基因nt.6295突变,导致ORF1ansp3的CDS(coding sequence),即SUD(SARS-CoV unique domain)出现了一个终止密码子,而后者可能参与调节病毒复制或抵抗受感染宿主的免疫反应。可见,此变异和病情轻,无续发有关。由此,Song等推断这次爆发是一次独立的(进化)事件。但我们不完全同意此观点。诚然爆发病情轻无续发与ORF1a基因nt.6295突变有关,而其并非孤立事件。这是SARS CoV进入人群后发生的一系列“逆向进化”过程之延续[1],故尚不能确定其为一次独立的(进化)事件,甚至很可能是全球SARS流行的收尾。
在流行病学上,分析流行或爆发原因或机制时很重视佐证,其作用犹如司法案件证据链中重要甚至关键环节。2004-03-25至4月末,我国突然发生一起实验室感染事件,在北京和安徽共确诊9例患者,死亡1例。后来也有续发和超级传播者:护理首例患者的一位护士传播5例(图3)[10-11]。可见其传播力强,流行强度大,病情危重,病死率(1/9)高。其流行病学特征和临床表现与同年1月广州爆发的SARS明显有别,却与2002~2003年的流行非常一致。从SARS流行过程看,这起实验室感染既非2002~2003年SARS流行之延续,也与之前3月发生的广州SARS爆发无关,应是独立的,因暴露于含SARS CoV之实验材料感染所致爆发。更有甚者,其分离的病毒和贮存于实验室内2003年上半年流行毒株的特性相同。可见,2003年底至2004年初人和狸群SARS CoV基因已“逆向进化”,所致病例症状轻,传播力弱,较2002~2003年已有明显区别[1];而这次实验室感染病例的流行病学与临床特点及其病毒特性却仍与2002~2003年相同,其惟一的原因是:致实验室爆发的SARS CoV贮存于实验室,未经人群传播中“逆向进化”。这又从另一方面证实,SARS CoV为非自然(基因改造)产生而进入人群后存在“逆向进化”。故这次实验室感染偶发事件不仅为解释全球SARS流行的整个过程,而且也为SARS CoV“逆向进化”提供了有力的佐证。
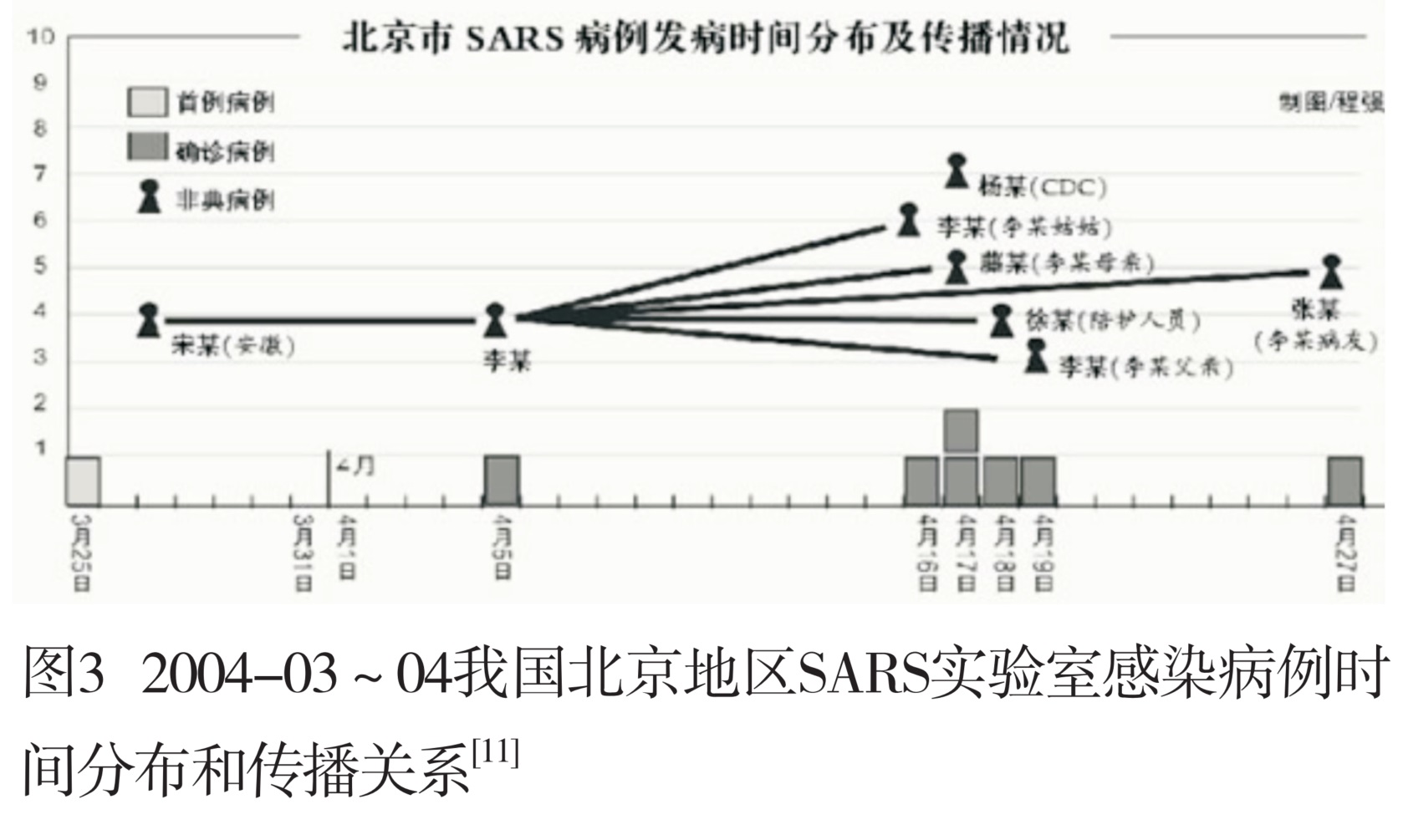
图3 2004-03~04我国北京地区SARS实验室感染病例时间分布和传播关系[11]
3、作为特定传染源(传播媒介动物)——受染果子狸分布非常奇特
传染病的分布为病原体及其进化、三环节和二因素相互作用所致。我们前面已述SARS整个流行过程分布异常情况、最早发病的地区广东分布异常情况、传播力和临床病情两极化演变异常情况以及实验室爆发佐证情况,现讨论三环节中第一、二环节即作为传染源和(或)传播媒介的动物分布异常情况。
自2003-05在果子狸等动物中分离到SARS CoV之后[12],起初很多学者相信果子狸是SARS的传染源(source of infection),甚至贮存宿主(reservoir),可能为整个流行的动物起源;继后许多事实又将其否定。这些研究者长期致力于在中国内地、中国香港和东南亚甚至其他洲的蝙蝠中寻找SARS CoV的起源和贮存宿主。然而,国内传染源被严格定义为:携带并能将病原体排出体外使易感者受染的人和动物。因此,受染的果子狸符合此定义。但实质上,从整个流行过程和果子狸分布来看,其和其他人兽共患病作为传染源的动物不同。果子狸仅在非常特定的状态,即市场饲养中且某一时段内才具传染源之作用。故为区别,我们暂称果子狸为SARS“特定传染源(peculiar source of infection)”。当然,若将果子狸作为SARS CoV传播媒介动物,应该是毫无疑问的,但其绝不是贮存宿主。在此,为定位果子狸在SARS流行中的作用,我们特别指出,对传染病流行病学理论的表述,在我国和西方有许多不同之处。如西方将传染源和贮存宿主混用,认为两者作用相同、定义一致,且更常用“reservoir”表述传染源,而我国将这两个名词做了明确区别,即贮存宿主为疫区内能世代保存该传染病病原体并维持传播与流行的动物和人。按此说来,贮存宿主为传染源,而传染源并非定为贮存宿主。因此,我国传染病和流行病学理论优于西方。事实上,我国能在解放后10多年时间内,取得防控甚至基本消除五大寄生虫病等流行广泛、危害极甚的重要传染病,上述理论的指导起到了关键作用。
国内外研究者对如此重要的传播环节进行了大量细致的调查研究工作,同时对我国内地携带SARS CoV和受染果子狸的分布亦做过许多研究,其中两次研究结果发表在国际杂志上[9,13-14]。两次的研究结果比较一致,并发现非常奇特的状态,即果子狸均集中于深圳和广州的活动物市场(live-animal market)或野生动物市场,且主要在广州新源动物(Xinyuan animal market)市场。Kan等[13]2004-01在新源市场内,从4个商贸区18个摊位随机抽取91只果子狸(Paguma larvata)、15只貉(Nyctereutes procyonoides)以及24份外环境标本,并于同年1~9月在国内和广东牲畜贸易大于80%的其他省份且供应新源动物市场的农场收集标本,一般每省两个,共计25个农场1107只果子狸。结果令人惊异,新源市场91只果子狸、15只貉SARS CoV均阳性;24份外环境标本中22份阳性;而其他省份1107个标本均阴性,地区分布结果竟如此截然不同。在论文报道中,也能找到此结论正确性的有力佐证。Kan等[13]发现一个农场主送至新源市场的17只果子狸,SARS CoV均阳性,而其位于河南省农场所有的果子狸(169只)均阴性。因而,学者们均认为,动物市场尤其是新源市场是SARS传染来源,而非果子狸饲养农场,即便是广东省内离广州很近的饲养场亦是如此。
遗憾的是,研究者再未进行深层次的思考:①为何和其他人兽共患病不同,动物传染源不是出于荒地、山林和农田等自然疫源地?②受染果子狸的来源均为动物市场,却非长期且群居的饲养农场?(有人指出这是因市场动物种类多且笼内密度高,但深思之,此理由难以完全解释。)③为何发现受染果子狸的动物市场仅为两个(深圳和广州新源市场)?④为何动物市场受染果子狸仅在2002~2004年发现?⑤为何深圳和广州两个动物市场的位置和上述第1点中流行早中期病例均在广州西面和南面的分布十分吻合(图2)?⑥此类动物市场是否仅深圳和广州有?仅中国大陆有?
可见,如此多的“特殊”、“为何”、“是否”等,应该考虑到公众和学者均不会也不想但现实又不得不面对的问题:SARS CoV与既往新发传染病尤其是人兽共患病有着异乎寻常的不同之处,即SARS CoV非自然地引入。在此值得提一事实,Xu等[6]进行流行病学研究时,发现我国广西的指示病例是位野生动物贸易商的司机,而这位贸易商将我国广西等其他省份甚至越南的野生动物供应到我国广东动物市场。由此可见,广东动物市场来往的渠道十分杂乱,甚至可以直通国外,是否可以推测这为“SARS CoV非自然地引入”提供了可乘之机。
综上所述,我们归结或强调两点:①我们通过比较人类传染病流行病学和临床的自然史,从3方面分析了SARS流行病学和临床特征的明显反常,为SARS CoV非自然(基因改造)起源之说提供了科学依据。有关SARS CoV基因突变与分子进化之逆向进化和其非自然(基因改造)起源的详细内容将另文发表。②SARS CoV非自然(基因改造)起源实际上意味着人类已进入经“新型人工病毒”致全球性流行的时代,故应群起应对,切勿掉以轻心。
致谢:本项目组成员王波副教授、谭雅慧硕士对本文也有贡献;本文得到本校陈景元、闫永平、夏洁来和陈长生教授、张景霞高级实验师的支持;笔者和陕西师范大学生命科学院黄原教授就生物进化的某些问题进行了讨论,受益匪浅;本校图书馆苏春萍副研究馆员和石建副研究馆员无私帮助收集文献;本校外语教研室樊家勇副教授对摘要的中译英给予重要指正。
【参考文献】
[1]徐德忠,孙慧敏,谭雅慧.现在自然界和人群中已不存在非典病毒[J]. 医学争鸣,2013,4(1):22-26.
[2]Song H,Tu C,Zhang G,et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human[J]. Proc Natl Acad Sci USA,2005,102(7):2430-2435.
[3]Hon CC,Lam TY,Shi ZL,et al. Evidence of the recombinant origin of a bat severe acute respiratory syndrome (SARS)-like coronavirus and its implications on the direct ancestor of SARS coronavirus[J]. Virol,2008,82(4):1819-1826.
[4]Balboni A,Battilani M,Prosperi S. The SARS-like coronaviruses:The role of bats and evolutionary relationships with SARS coronavirus[J]. New Microbiol,2012,35(1):1-16.
[5]Chinese SARS Molecular Epidemiology Consortium.Molecular evolution of the SARS coronavirus during the course of the SARS epidemic in China[J]. Science,2004,303(4):1666-1669.
[6]Xu RH,He JF,Evans MR,et al. Epidemiologic clues to SARS origin in China[J]. Emerg Infect Dis, 2004,10(6):1030-1037.
[7]罗会明 ,黄吉城 ,周端华,等.广东省2003~2004年SARS的流行与控制[J].中国人兽共患病学报,2006,22 (4):326-329.
[8]Liang GD,Chen QX,Xu JG,et al. Laboratory diagnosis of four recent sporadic cases of community-acquired SARS,Guangdong Province,China[J]. Emerg Infect Dis,2004,10(10):1774-1781.
[9]Tu C,Crameri G,Kong X,et al. Antibodies to SARS coronavirus in civets[J].Emerg Infect Dis, 2004,10(12):2244-2248.
[10]张朝武,姚玉红,王国庆.从SARS-CoV实验室感染看生物安全的重要性[J].现代预防医学, 2004,31(5):656-660.
[11]北京发布4月非典疫情流行控制情况分析报告(图).新京报. http://www.sina.com.cn, 2004-07-01,06:24.
[12]Guan Y,Zheng BJ,He YQ,et al. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China[J]. Science,2003,302(6):276-278.
[13]Kan B,Wang M,Jing H,et al. Molecular evolution analysis and geographic investigation of severe acute respiratory syndrome coronavirus-like virus in palm civets at an animal market and on farms[J]. J Virol,2005,79(18):11892-11900.
[14]Martina BE,Haagmans BL,Kuiken T,et al. Virology:SARS virus infection of cats and ferrets[J]. Nature,2003,425(12):915.
相关阅读二:逆向进化:SARS CoV非自然起源之关键
作者:徐德忠 张景霞 张 磊 孙慧敏 来源:《医学争鸣》2014年第1期
我们已分别从概况和流行病学反常分布等方面阐明SARS CoV非自然起源的理由[1-2]。本文将描述其系统发育和“逆向进化”;正由此使我们考虑到,其直接祖先为非自然进化而来。因此,①揭示2003年底至2004年初广州爆发SARS CoV与2002〜2003年流行早期病毒之亲缘较晚期更近以及流行中其阳性选择压逐渐下降;②揭示其受体结合位点重要氨基酸的变异及其逆向进化;③揭示其和Bt-SLCoV(蝠SARS样冠状病毒)基因ORF8特征性29-nt之差异及其缺失和逆向进化;④揭示其基因ORF1ansp3的CDS(编码序列)(SUD:SARS CoV特有CDS)之变异并呈逆向进化趋势。这些论述十分重要,为证明其非自然起源之关键。
1、生物系统发育中的逆向进化
Bull最早定义逆向进化:“相同特征的分枝群体,经包括适应在内的方式,再获得祖先群体的特征[3]”;Teotcfnio[4]精简为“再获得祖先状态”。实际上,逆向进化在生物界随处可见,但除少数涉及人类生活事件外,不易引起人们的注意[3-5]。由于条件受限等,目前多见其实验和理论研究,尤其是对微生物、原生动物和昆虫[3-8]。但也有和人类环境密切相关的广泛研究个例:19世纪欧美工业空气污染和20世纪治理引起的胡椒蛾桦尺蠖由淡色变黑又变回的逆向进化,研究已进行几十年,最近仍有报道[6-8]。我们认为,逆向和顺向进化共同形成了生物进化。
中医阴阳学说认为,世界包括人体健康和疾病、人群发病和流行均在阴阳二气相互作用下发生发展,二者既相互对立又相互统一[9]。可见,逆向进化也符合哲学和医学的基本理论。
2、 SARS CoV 经历的逆向进化
以此为指导,我们重新研究、比较分析了国内外的大量工作[10-29]。虽然学者们对结果做了深人的常规分析,有具体的新发现,但对于SARS CoV跨种的确切机制和贮存宿主,均言:“需进一步研究”;对其直接祖先,同声:“应全面搜索”。而本文以另一角度揭示了其逆向进化之规律,为探讨真实起源打下科学基础。
2.1 2003~2004年广州爆发SARS CoV与2002〜2003年流行早期病毒之亲缘较晚期更近以及流行中其阳性选择压逐渐下降
国际上将2002~2003年流行分成早、中和晚期[1,2,19,29]。关于“2003~2004年广州爆发SARS CoV与2002~2003年流行早期病毒之亲缘较晚期更近”,已报道[1]。
适应性进化通常以非同义置换速率与同义置换速率比值(Ka/Ks)(theratiosoftheratesofnonsynonymoustosynonymous)衡量。若Ka/Ks>1,良P具阳性选择压;若Ka/Ks<1,即处净化选择,不适应的序列将被淘汰。学者们早在《Science》杂志上,分析了SARS CoV进化过程,从早经中至晚期,其Ka/Ks总体上逐渐下降。两两分析显示:①S和ORF1a基因类似:初受强烈阳性选择压力,后为净化选择并最终稳定;②而ORFlb流行全过程似均经历净化选择;但该序列为基因组最保守的区域。此令人深省!该文作者特别指出:这是一非凡的进化历程,但其机制需进一步研究[29]。
在此,结合SARS CoV根本不存在贮存宿主和其流行后已从自然界与人群消失之现实,本文应用此理论,解释上述选择性适应和净化过程所致非凡进化历程的机制:①由于ORF1b为病毒基因组最保守区域,在正常进化中,其序列被淘汰的概率应远低于S等基因,但实际却相反,为进化史之反常;②ORF1b序列的某些核苷酸被淘汰,从流行病学理论判断:其不适应人类;③最保守序列不适应人类,原因可能仅一个:其经历了“非自然进化”[1],即基因改造;④SARS CoV进人人类后,必遭强大压力,必以逆向进化应对,表现为全过程经历净化选择。《Science》之问由此迎刃而解。
2.2 SARS CoV受体结合位点重要氨基酸出现逆向进化
目前公认,Bt-SLCoV是SARS CoV隔数代的共同祖先,尤其是Rp3株(亲代3)[20-21,30]。我们研究SARS CoV受体结合位点重要氨基酸(AA)时发现,和SARS CoV传播力密切相关、与患者(人)或果子狸(狸)受体血管紧张素转换酶2(ACE2)之结合位点为S1318-510位点[16,19-20,22-24,31]。其中,487、479两位AA尤为重要;已用突变、变异和比对、免疫试验、嵌合ACE2和X射线晶体学等实验进行了验证。Li等指出487、479两位AA之作用:2002~2003年流行中若变异,如(N479K)增加电荷和关键甲基基团(T487S)缺失,即像2003~2004年广州爆发人和狸株那样,则其和ACE2的亲和力差1000倍以上[23-26]。其宗旨为寻找跨种传播的突变热点;由于未用甚至未知“逆向进化”理论,对起源仍仅寄托于“未来研究”,但却在客观上为我们逆向进化的研究提供了依据。
在此,我们将487、479两位AA,从祖先Bt-SLCoV开始,经2002~2003年流行至2003~2004年广州爆发狸和人SARS CoV的变异历程进行综合分析(表1)。发现:479位AA:在Bt-SLCoV(亲代3)为S(丝氨酸);至2002~2003年流行时狸株,为另3种同时存在:R(精氨酸)、K(赖氨酸)、N(天冬酰胺);而人株,仅1种:N。令人惊异:2003-2004年广州爆发时狸株仅为原先的2种:R、N;人株却增至3种,和2002-2003年流行时狸株相同:R、K和N。487位AA也类似:Bt-SLCoV为V(缬氨酸);至2002~2003年流行时狸株为S;人株为T(苏氨酸)。同样令人惊异:2003~2004年广州爆发时人和狸同,均为S。

很显然,两位AA从祖先开始,至广州爆发止,其进化经一或多种不完全的历史回复:蝠株(亲代3)---?(亲代2)---?(亲代1)---2002~2003年狸株---2002-2003年人株---2003-2004年狸株---2003-2004年人株[1]。该过程很可能更复杂曲折。然而,方向仅一个:2003〜2004年人株向2002-2003年狸株回复,再结合前文广州爆发病例的临床和流行病学特点[1-2],不难推断SARS CoV经历了逆向进化。Janies等[31]专门构建了不同宿主SARS CoV或Bt-SLCoV487、479位AA的系统发育树。从中,可更清楚地了解我们的观点。
3、 SARS CoV ORF8特征性29-nt存在逆向进化
人和狸SARS CoV基因组,除前述的主要基因外,具独特的附属基因,即ORF8,后者含特征性29-nt序列:CCTACTGGTTACCAACCTGAATGGAATAT(nt.27869-27897)。早在2003年学者发现,除2002-2003年流行早期极少数患者外,特征性29-nt在大部分病例中缺失。故将此谓特征性29-nt缺失,并认为其和SARS CoV从动物至人类的适应性有关[32]。此后研究[19,29-31,33-35],证实其可能与SARS CoV复制、致病性与跨种传播等有关。鉴于此,我国学者发表文章,特别画出ORF7b-ORF8之RNA二级结构,且将GZ02(存在29-nt)和TOR2(缺失)2株病毒进行比较[29]。
无独有偶,Bt-SLCoV同样存在ORF8特征性29-nt序列:CCAATACATTACTATTCGGACTGGTTTAT(nt.27866-27894),且①仅在蝠内,为其特有;②和宿主蝠联系紧密;③和人-狸29-nt序列相比,位点提前3位,呈直系基因位置关系,有12位nt呈多态性。为易表述和区别,我们谓其“原29-nt”;人-狸nt.27869-27897,称为“变29-nt”。我们进一步发现,如同前述487、479两位AA—样,ORF829-nt在进化历程中,有十分类似的逆向进化(表1)。狸株均为“变29-nt”,但人株,除2002~2003年流行最早期少数外,绝大部分均缺失;而至2003~2004年广州爆发,却又回复“变29-nt”,如同狸株,呈现典型的逆向进化。
上述3点,仅为我们发现的主要逆向进化,但肯定还有更多未知数。如上述,SARS CoV或某些Bt-SLCoV有一独特的CDS,即SUD[14,19,36]。SUD具357个AA,3个亚结构域;其参与调节病毒复制或抵抗受感染宿主的免疫反应。Song等[19]指出,2003~2004年广州爆发人株的ORF1ant.6295突变,导致SUD出现终止密码子;并认为其和病例的症状轻与传染性低有关。又如前述,他们的目标欲证明:广州爆发为区别于2002~2003年流行的独立事件,但客观上却提供了逆向进化又一证据。因SUD功能减弱或丧失,反映SARS CoV不能适应人群,发生回复变化。
但需指出,SARS CoV逆向进化的方式和以往有非常明显的差异,甚至史无前例。因为:①SARS CoV的进化并非完全在生物界和人类中进行;②其曾经非自然进化处理;③其寿命之短,在同类病毒甚至病毒界,可能绝无仅有;④人群流行中,其毒力和传播力变异相当悬殊:强时致病8098例,亡774例,现有许多超级传播者,波及全球;弱时仅一城市致4例,无续发;两者之间时隔仅半年,为生物界传染病史之一瞬;但实验室毒株的毒力和传播力却未变。到此,应该回答下一问题:SARS CoV为何出现逆向进化,又如何与其起源和消失有关?
4、 SARS CoV 逆向进化的原因及其起源与消失之框架
虽逆向进化是生物进化中的正常过程,普遍存在,但我们对其知之甚少。超乎常识,逆向进化可呈顽强的表现,如估计,鲸类后肢返祖的能力约可保留107代,但不可逆性仅存很短时间[4,37]。所以,SARS CoV呈现逆向进化不足为奇。
逆向进化的直接原因很复杂,主要为生存环境发生了变化。我们归纳,可能有3点:①若群体迁人新环境时间很短,其易保持祖先环境产生的永久遗传变异;②返回祖先状态时,基因型中有利于祖先环境变异的频率增加;③新环境中,新的选择变异株少,新选择性差,利于逆向进化。
为解答SARS CoV逆向进化的具体原因,必须回顾其进化和所致流行之要点〜2]:①其最早祖先可能为Bt-SLCoV,共同祖先时间约在爆发前4.5年,而HIV从源病毒至人,约经700~800年;②至今未发现其直接祖先和贮存宿主;③2002~2003年流行仅持续8~9个月,并无疫苗预防,且爆发和消失的时间点均突然;④其和其他CoV所致病的流行病学及临床特点相去甚远;⑤其和新发传染病的病毒进化与流行之普遍规律不符;⑥其呈现许多逆向进化;⑦流行后10年再未见病例和动物受染。至此,一条证据链形成:SARS CoV是“非自然”所致,即由Rp3株或类似毒株经非自然改造所致,否则不能解释上述诸点。
因此,进人新宿主后,不断受到强大逆向进化之压力。由于:①离祖先病毒株仅约4年多时间,故其产生后,不可能在人类亲缘很近的动物群内做适应试验;②同理,迅即进人全新环境,易保持祖先环境的永久遗传变异;③新宿主中,其新的选择条件差,未见许多新亚型;而阳性选择压不断降低,利于祖先环境变异的频率增加。所以,其进人人群后,特征性29-nt序列迅速丢失;现一系列逆向进化;一年后毒力和传播力明显下降,不久即在人类和自然界消失。这应是其最好的归宿。
根据上述的论证,我们构建了“SARS CoV的起源和进化”框架图(图1)。
关于图1,还需强调一要点,即非自然(基因)改造技术及其应用。虽然我们均已描写[1-2],但很多读者、甚至学者对此仍未完全了然。图1个别环节还需完善和修改,但在总体上可合理地解释以上疑问和事实。

①图中框分二层:上层为SARSCoV的起源和进化流程,下层除“实验室感染”外,为上层框内容之描述;②487、479:SARSCoV或Bt-SLCoVS1区487、479两位AA,数字前或后的英文为AA之种类;③原29-nt,变29-nt,无29-nt:分别代表其特征性29-nt序列:CCAATACATTACTATTCGGACTGGTTTAT(nt.27866-27894),CCTACTGGTTACCAACCTGAATGGAATAT(nt.27869-27897)和缺失;④SUD,终码SUD:分别代表nt.6295突变出现终止密码子前后的SUD;⑤毒株:早GZ02GD01:2002~2003年流行早期2人株;晚Tor2:晚期人株;狸SZ3:早期1狸株;2004年GZ04:2003~2004年广州爆发人株;PC04:为其狸株。特别说明:这些毒株应看作为该期毒株的代表;⑥实验室感染。其次,目前自然界己不存在SARSCoV,仅贮存实验室。
图1“SARSCoV的起源和进化”框架图
本文可得如下结论:①在人类动物源性传染病发展史上,我们首次提出由非完全动物源性导致该病全球流行之可能;②人类实际已进人利用非自然(基因)改造技术产生新病毒并使之在人群中流行的时代,应引起WHO和全世界的共同关注;③逆向进化理论在指导人们认识传染病起源方面可发挥重要作用。
由于事关人类健康和全球安全,通过本文之研究,我们专业学者有责任、有义务,再次吁请我国卫生和计划生育委员会正式向WHO提出建议:组织一个专家委员会,和有关国家合作,完成两项任务:①对人群和动物群体进行调查研究,实证SARS CoV已经从中消失,并正式宣布:人类历史上已消灭了第二种传染病——SARS,以使公众安心;也慰籍无私奉献、专业超群的科学家Dr.CarloUrbani,他是判定SARS爆发的首位WHO官员,后在岗位上受染而牺牲[11,38]②在专门实验室,验证某些必要的非自然(基因)改造技术。
致谢:我校陈景元、闫永平、夏洁来和陈长生教授,王波和樊家勇副教授,苏春萍副研究馆员的支持或帮助。
[参考文献]
[1] 徐德忠,孙慧敏,谭雅慧.现在自然界和人群中已不存在非典病毒[J].医学争鸣,2013,4(1):22-26.
[2] 徐德忠,张磊,孙慧敏,等.SARS CoV非自然起源[J].医学争鸣,2013,4(4):6-9.
[3] Bull JJ,Charnov EL. On irreversible evolution[J]. Evolution, 1985,39(5): 1149-1155.
[4] TeotOnio H,Rose MR. Perspective: reverse evolution[J]. Evolution,2001,55(4):653-660.
[5] Desai MM. Reverse evolution and evolutionary memory[J].Nat Genet,2009,41(2):142-143.
[6] Cook LM,Grant BS. Frequency of insularia during the decline in melanics in the peppered moth Biston betularia in Britain[J].Heredity (Edinb),2000,85(Pt 6):580-585.
[7] Grant BS,Wiseman LL. Recent history of melanism in American peppered moths[J]. J Hered, 2002, 93(2):86-90.
[8] Saenko SV, JerOnimo MA, Beldade P. Genetic basis of stage-specific melanism: a putative role for a cysteine sulfinic acid decarboxylase in insect pigmentation [J].Heredity (Edinb),2012,108(6):594-601.
[9] 郑守曾.中医学第5版.北京:人民卫生出版社,1999:26-43.
[10] Marra MA,Jones SJ,Astell CR,et al. The genome sequence of the SARS-associated coronavirus[J]. Science, 2003, 300(5624):1399-1404.
[11] Ksiazek T,Erdman D,Goldsmith C,et al. A novel coronavirus associated with severe acute respiratory syndrome[J]. N Engl J Med, 2003, 348(20):1953-1966.
[12] Rota PA,Oberste MS,Monroe SS,et al. Characterization of a novel coronavirus associated with severe acute respiratory syndrome[J]. Science,2003,300(5624):1394-1399.
[13] Ruan YJ,Wei CL,Ee AL,et al. Comparative full-length genome sequence analysis of 14 SARS coronavirus isolates and common mutations associated with putative origins of infection[J]. Lancet,2003,361(9371):1779-1785.
[14] Snijder EJ,Bredenbeek PJ,Dobbe JC,et al. Unique and conserved features of genome and proteome of SARS- coronavirus,an early split-off from the coronavirus group 2 lineage[J]. J Mol Biol,2003,331(5):991-1004.
[15] Guan Y,Zheng BJ,He YQ,et al. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China[J]. Science,2003,302(5643):276-278.
[16] Kan B,Wang M,Jing H,et al. Molecular evolution analysis and geographic investigation of severe acute respiratory syndrome coronavirus-like virus in palm civets at an animal market and on farms[J]. J Virol,2005,79(18):11892-11900.
[17] Wang M,Yan M,Xu H,et al. SARS-CoV infection in a restaurant from palm civet[J]. Emerg Infect Dis,2005, 11(12):1860-1865.
[18] Xu RH,He JF,Evans MR,et al. Epidemiologic clues to SARS origin in China[J]. Emerg Infect Dis,2004,10(6):1030-1037.
[19] Song H,Tu C,Zhang G,et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human[J]. Proc Natl Acad Sci U S A,2005,102(7): 2430-2435.
[20] Yip CW, Hon CC, Shi M, et al. Phylogenetic perspectives on the epidemiology and origins of SARS and SARS-like coronaviruses[J]. Infect Genet Evolut,2009,9(6):1185-1196.
[21] Hon CC,Lam TY,Shi ZL,et al. Evidence of the recombinant origin of a bat severe acute respiratory syndrome (SARS)- like coronavirus and its implications on the direct ancestor of SARS coronavirus[J]. J Virol,2008,82(4):1819-1826.
[22] Balboni A, Battilani M, Prosperi S. The SARS-like coronaviruses: the role of bats and evolutionary relationships with SARS coronavirus[J]. New Microbiol,2012,35(1):1-16.
[23] Li F, Li W, Farzan M, et al. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor[J]. Science,2005,309 (5742):1864-1868.
[24] Holmes KV. Structural biology. Adaptation of SARS coronavirus to humans[J]. Science,2005,309(5742):1822-1823.
[25] Li F. Structural analysis of major species barriers between humans and palm civets for severe acute respiratory syndrome coronavirus infections[J]. J Virol,2008,82(14): 6984-6991.
[26] Wu K, Peng G, Wilken M, et al. Mechanisms of host receptor adaptation by severe acute respiratory syndrome coronavirus[J].J Biol Chem,2012,287(12):8904-8911.
[27] Adedeji AO,Marchand B,Te Velthuis AJ,et al. Mechanism of nucleic acid unwinding by SARS-CoV helicase[J]. PLoS One,2012,7(5):e36521.
[28] Yu MS, Lee J, Lee JM, et al. Identification of myricetin and scutellarein as novel chemical inhibitors of the SARS coronavirus helicase,nsP13[J]. Bioorg Med Chem Lett,2012, 22(12):4049-4054.
[29] Chinese SARS Molecular Epidemiology Consortium. Molecular evolution of the SARS coronavirus during the course of the SARS epidemic in China[J]. Science, 2004, 303(5664):1666-1669.
[30] Li W,Shi Z,Yu M,et al. Bats are natural reservoirs of SARS-like coronaviruses[J]. Science,2005,310(5748):676-679.
[31] Janies D,Habib F,Alexandrov B,et al. Evolution of genomes, host shifts and the geographic spread of SARS-CoV and related coronaviruses[J]. Cladistics,2008,24(2):111-130.
[32] Enserink M. Clues to the animal origins of SARS[J]. Science, 2003,300(5624):1351.
[33] Wu D, Tu C, Xin C, et al. Civets are equally susceptible to experimental infection by two different severe acute respiratory syndrome coronavirus isolates[J]. J Virol,2005, 79 (4): 2620-2625.
[34] Qin E, He X, Tian W, et al. A genome sequence of novel SARS-CoV isolates: the genotype, GD-Ins29, leads to a hypothesis of viral transmission in South China[J]. Genomics Proteomics Bioinformatics,2003,1(2):101-107.
[35] Oostra M, de Haan CA, Rottier PJ. The 29-nucleotide deletion present in human but not in animal severe acute respiratory syndrome coronaviruses disrupts the functional expression of open reading frame 8[J]. J Virol,2007,81(24): 13876-13888.
[36] Johnson MA, Chatterjee A, Neuman BW, et al. SARS coronavirus unique domain: three-domain molecular architecture in solution and RNA binding[J]. J Mol Biol,2010,400(4):724-742.
[37] Castro F, Gaedke U, Boenigk J. Reverse evolution-driving forces behind the loss of acquired photosynthetic traits[J].PLoS One,2009,4(12):e8465.
[38] World Health Organization. Dr. Carlo Urbani of the World Health Organization dies of SARS[EB/OL]. (2003-03-29).http://www.who.int/csr/sars/urbani/en/.